La resistencia de Leishmania al antimoniato de meglumina o Glucantime® es uno de los principales fenómenos evidenciados en los pacientes con falla terapéutica, cuyo estudio es imprescindible para el diseño de nuevas alternativas terapéuticas. En este trabajo se reportan los resultados de un estudio comparativo del perfil genético de cepas resistentes y no resistentes al Glucantime®, con el objetivo de identificar posibles marcadores genéticos relacionados con la resistencia al fármaco en los casos de leishmaniasis cutánea localizada. Para ello, se realizó inducción de resistencia in vivo e in vitro al fármaco en cepa de referencia internacional de L. (V.) braziliensis y se comparó con aislados provenientes de pacientes con o sin falla terapéutica. Se realizó digestión con enzimas de restricción (HindIII, PstI, MspI) y electroforesis en gel de agarosa al 1%, haciendo uso de comparación manual y estadística descriptiva para el análisis. Se evidenciaron bandas de mayor intensidad, con peso molecular entre 2,0 y 1,5 kb en uno de los aislados sin curación clínica (HindIII), y dos bandas distintivas de 0,65 kb y 0,5 kb (MspI) en el aislado con inducción de resistencia experimental. La mayoría de los pacientes informó como procedencia y sitio de probable infección al estado Miranda (57,14% y 71,42%, respectivamente), y una minoría (28,58%) tuvo falla terapéutica. Fue posible identificar variaciones en los perfiles de restricción de aislados resistentes experimentalmente inducidos y de pacientes con falla terapéutica. Adicionalmente, se propone un nuevo método para la inducción de resistencia al fármaco en condiciones in vivo e in vitro.
Palabras clave: Leishmaniasis; Tratamiento; Resistencia; Marcadores moleculares; Antimoniato de Meglumina.
Leishmania resistance to meglumine antimoniate or Glucantime® is one of the main phenomena evident in patients with therapeutic failure, the study of this phenomena is essential for the design of new therapeutic alternatives. In this work we describe an experimental study carried out in order to do a comparative analysis of the genetic profile of resistant and non-resistant strains to Glucantime®, and the identification of possible genetic markers related to drug resistance in cases of localized cutaneous leishmaniasis. To do this, induction of resistance in vivo and in vitro to the drug was performed using the therapeutic dose of glucantime with the international reference strain of L. (V.) braziliensis and compared with samples from patients with or without therapeutic failure. Digestion with restriction enzymes (HindIII, PstI, MspI) and 1% agarose gel electrophoresis were performed, using manual comparison and descriptive statistics for the analysis. Bands of greater intensity were evident, with a molecular weight between 2.0 and 1.5 kb in one of the patients without clinical cure (HindIII), and two distinctive bands of 0.65 kb and 0.5 kb (MspI) in the isolated with induction of resistance experimentally. The majority of the patients came from Miranda state and it is the probably site of infection (57.14% and 71.42%, respectively), and a minority (28.58%) had a therapeutic failure. It was possible to identify variations in the restriction profiles of experimentally resistant parasites and that from patients with therapeutic failure. In addition, a new method for the induction of drug resistance under in vivo and in vitro conditions was proposed.
Key words: Leishmaniasis; Treatment; Resistance; Molecular Markers; Meglumine Antimoniate.
La leishmaniasis es una enfermedad de transmisión vectorial, causada por parásitos del género Leishmania spp, la cual se encuentra ampliamente extendida en todo el mundo, especialmente en regiones tropicales y en pacientes con estrato socioeconómico bajo (Handler et al., 2015). Desde que un conjunto de científicos (como Leishman, Donovan y Viannia) descubrieron, de forma independiente, el microorganismo causante de la enfermedad, numerosas investigaciones han sido llevadas a cabo para caracterizar a nivel básico y clínico una de las parasitosis más frecuentes en la actualidad, después de la malaria o paludismo (OMS, 2010).
Las opciones terapéuticas de la enfermedad incluyen drogas convencionales como los antimoniales pentavalentes (SbV), Anfotericina B, Miltefosina, Pentamidina y Paramomicina (De Guglielmo et al., 2018). Los primeros (SbV) incluyen a su vez, el antimoniato de meglumina o Glucantime® (GLU) y el Estibogluconato de sodio, los más frecuentemente usados en Latinoamérica, según los datos disponibles para el año 2008 (Tuon et al., 2008), logrando la cura satisfactoria del 76,5% de los casos. En este orden de ideas, a pesar de que el tratamiento con Pentamidina ha mostrado resultados relativamente similares; la experiencia terapéutica, el costo y los efectos adversos son factores que condicionan a los SbV como la primera opción terapéutica de la región.
Sin embargo, la resistencia adquirida al GLU (Berg et al., 2013) es un fenómeno cuya frecuencia ha incrementado con el paso de los años, inicialmente en India (Lira et al., 1999), lo cual obligó a las autoridades competentes a la aplicación de alternativas terapéuticas. A partir de ese momento, la resistencia a los SbV se ha reportado en otras regiones. Este fenómeno puede estudiarse a nivel fenotípico, proteómico o genómico (Jeddi et al., 2011), siendo este último evaluado, entre otros métodos, mediante el análisis de longitud de fragmentos de restricción con endonucleasas, el cual permite evaluar la variabilidad genética entre aislados resistentes y sensibles al tratamiento, mediante la comparación de los patrones o perfiles de restricción obtenidos.
La falla terapéutica con los fármacos convencionales es un fenómeno multifactorial, y depende en buena medida de factores relacionados al hospedador, coinfecciones, al fármaco y al parásito (Ponte-Sucre et al., 2017; Aït-Oudhia et al., 2011). Dentro de este último grupo de factores, se incluye la resistencia al tratamiento (innata o adquirida), reportada inicialmente en India (Lira et al., 1999) y extendida eventualmente a otras regiones del mundo mediante estudios de susceptibilidad a antimicrobianos (Sereno et al., 2019). Sin embargo, los datos epidemiológicos sobre dicha problemática son escasos, por lo que la necesidad de conocer el estado actual de resistencia en los países en los que los antimoniales pentavalentes siguen siendo la primera opción terapéutica, son estrictamente necesarios.
El objetivo del presente trabajo fue realizar un análisis comparativo entre el perfil genético observado en cepas experimentalmente resistentes y no resistentes al GLU, además de muestras provenientes de pacientes con y sin falla terapéutica, con la finalidad de identificar posibles marcadores genéticos relacionados con la resistencia al fármaco en casos de leishmaniasis cutánea localizada.
Se realizó un estudio experimental, cuyos aspectos metodológicos básicos para la obtención de muestras y la posterior comparación de perfiles genéticos se resumieron en el algoritmo correspondiente a la figura 1.
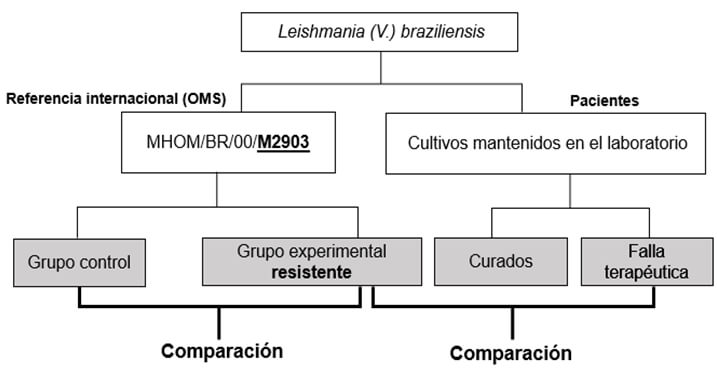
Para la inducción de resistencia in vivo al GLU, se utilizó la cepa de referencia internacional de Leishmania (V) braziliensis (MHOM/BR/00/M2903).
Además, se seleccionaron cultivos de parásitos previamente extraídos de pacientes que acudieron a la consulta de dermatología del IBJC. En ambos casos, los aislados se encontraban en el banco de muestras biológicas del laboratorio de ingeniería genética del IBJC.
El proceso de selección de muestras provenientes de pacientes se realizó mediante muestreo no probabilístico por conveniencia, según disponibilidad de las mismas en el laboratorio y con base en los criterios de inclusión en el estudio. Estos últimos consistieron en poseer, según los datos disponibles en la historia clínica del paciente de origen: (a) diagnóstico clínico de leishmaniasis cutánea localizada, (b) tratamiento con antimoniato de meglumina (Glucantime®) intralesional a dosis terapéuticas (10-20 mg/kg/día) con duración según criterio clínico y (c) determinación de especie por métodos moleculares compatibles con infección por Leishmania (V.) braziliensis. Los criterios de exclusión incluyeron: (a) contaminación de la muestra y (b) mala calidad del material genético extraído.
Estos se estratificaron según la respuesta al tratamiento como “curados” y “no curados”, con base en los criterios planteados por la OMS (PAHO, 2018). Adicionalmente, y con fines descriptivos, se identificaron las siguientes variables: duración del tratamiento hasta lograr la cura clínica, edad, sexo, procedencia y lugar probable de infección.
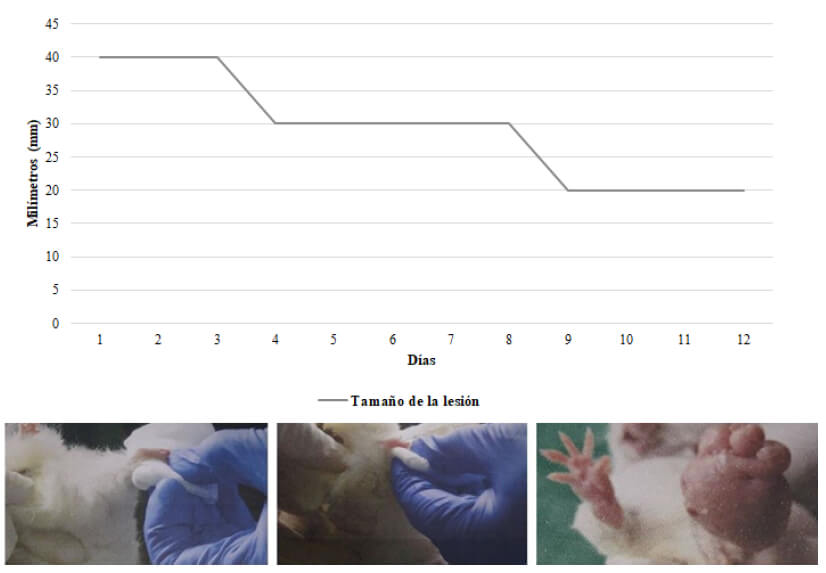
La cepa de referencia de Leishmania (V.) braziliensis (MHOM/BR/00/M2903) fue inoculada en la almohadilla plantar de una de las patas traseras de ratones de la cepa BALB/c, posterior a lo cual se esperaron ocho semanas antes de obtener una lesión significativa para comenzar el tratamiento intralesional. Dicho procedimiento consistió en pesar diariamente al animal, así como también medir el tamaño de la pata lesionada y la sana para determinar el tamaño de la lesión. Se administró una dosis diaria de antimoniato de meglumina de 20 mg/kg durante 12 días. Cuando se cumplió el tratamiento, se procedió a tomar una biopsia de la lesión para la obtención de los parásitos. Los mismos se mantuvieron en medios de cultivo con la presencia de GLU, con el objetivo de mantener la presión selectiva del fármaco.
Los parásitos aislados se mantuvieron en medio líquido M199 (Difco®) suplementado con suero fetal bovino (al 10%), penicilina (10,000 U/mL), estreptomicina (10,000 μg/mL) y GLU (4 mg/mL), realizando pases sucesivos semanalmente. Después de la inducción de resistencia al fármaco, los mismos fueron cultivados masivamente y cuando el crecimiento del grupo resistente (determinado por recuento en cámara de Newbauer) bajo la presencia de GLU fue igual al de la cepa sensible (en un medio de cultivo distinto sin GLU), se procedió a la extracción de ADN total (ADNt), al quinto día de cultivo. Los parásitos considerados resistentes se denominaron “grupo experimental”, mientras que aquellos que no estuvieron expuestos al medicamento fueron denominados “grupo control”.
Para la extracción y cuantificación de ADNt se realizó el mismo procedimiento realizado en otro estudio (Rodríguez et al., 2002). En cuanto a la determinación de especie por PCR, por cada aislado proveniente de paciente, se realizó el procedimiento con 100 ng de ADNt en una mezcla de reacción que contuvo 200 ng del oligonucleótido B1 (5’-GGG GTT GGT GTA ATA TAG TGG-3’) y B2 (5’-CTA ATT GTG CAC GGG GAG G-3’); 2,5 mM de deoxinucleotidos (dNTPs) y 2,5U de la enzima Taq Polimerasa. La mezcla de reacción fue sometida a 35 ciclos de amplificación (luego de 10 minutos a 90 °C para lograr la completa desnaturalización). Cada ciclo consistió en 1 minuto a 72 °C para amplificación.
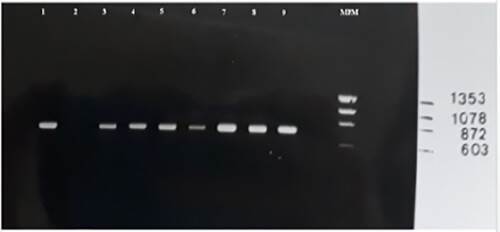
Para considerar a una muestra positiva para L. (V.) braziliensis, se debía evidenciar el producto esperado de 750 pb.
Se utilizaron las endonucleasas de restricción HindIII, PstI y MspI (Promega®, 10 U/μL), mezclando 10 μg de ADNt, 1 μL de enzima, 5 μL del buffer de la enzima (10X) y agua destilada (hasta alcanzar un volumen de 20 μL). Las mezclas se incubaron a 37°C durante 24 horas. Se realizaron electroforesis en gel de agarosa al 1% con buffer TBE y tinción con 5 μL de bromuro de etidio (0,5 μg/μL) para la visualización con luz ultravioleta, tomándose una fotografía de cada gel (MultiDoc-It UV Transiluminator de Analytic Jena®).
Para el análisis se usó estadística descriptiva. Las variables cuantitativas se expresaron con medidas de frecuencia central (media), y las cualitativas en forma de proporciones como porcentajes. El análisis comparativo de las bandas observadas se realizó de forma manual.
Se aplicó una inyección intralesional diaria del fármaco según dosis ponderal previamente establecida, durante 12 días utilizándose 15 μL del fármaco diariamente, no se registraron cambios de peso durante dicho período. En total, se administraron 216 μL del fármaco durante los 12 días de tratamiento. En la figura 2 se observa de forma gráfica la disminución progresiva de la lesión en la almohadilla plantar trasera del ratón, determinada por la diferencia de tamaño diario entre la pata sana y la lesionada.
Se seleccionaron 7 muestras provenientes de pacientes que cumplieron con los criterios de inclusión en el estudio. El promedio de edad de los pacientes al momento del diagnóstico fue de 44 años, existiendo ligera tendencia hacia el sexo femenino (57,14%) frente al masculino (42,86%). En orden decreciente de frecuencia, los pacientes provinieron del estado Miranda (57,14%), Vargas (28,57%) y el Distrito Capital (14,29%). En cuanto al posible lugar de infección, predominó el estado Miranda (71,42%), seguido de los estados Vargas y Táchira, con la misma proporción cada uno (14,29%). La mayoría de los pacientes (71,42%) fueron calificados como “curados” y el resto (28,58%) como “no curados”. El promedio de duración del tratamiento fue de 4,8 y 13 meses para cada grupo, respectivamente.
Se realizó la PCR según el procedimiento antes descrito para la determinación de especie, obteniéndose el producto esperado de 750 pb, tal como se muestra en la figura 3.
Luego de la extracción de ADNt y cuantificación del material genético, se procedió a realizar las digestiones con endonucleasas de restricción, que se realizaron según el protocolo antes descrito. En la figura 4, se observa una electroforesis en gel de agarosa que muestra el material genético proveniente de las muestras aisladas de pacientes con la enzima HindIII. Al comparar las muestras, es posible evidenciar diferencias significativas dadas por bandas de mayor intensidad correspondiente a las bandas entre 2 y 1,5 kb en la muestra 1 de paciente (P1) respecto a la muestra 3 (P3).
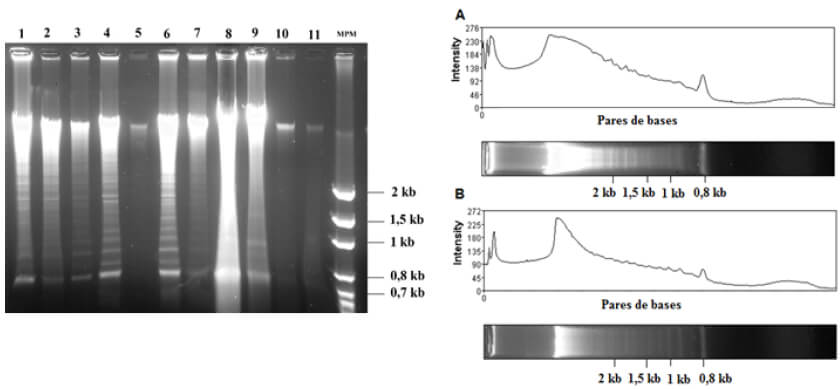
En la figura 5 se observa una comparación entre una muestra de un paciente con falla terapéutica (P1) y el del grupo control y experimental, digeridos con la enzima PstI y MspI, por separado. En el grupo control se evidencia una banda de aproximadamente 0,6 kb. En el aislado resistente se puede apreciar alrededor de los 0,65 kb, por último, una banda de casi 0,7 kb correspondiente a P1. Por lo tanto, se identificaron diferencias sutiles en cada uno de los aislados, obteniendo un perfil de restricción distinto en cada caso.
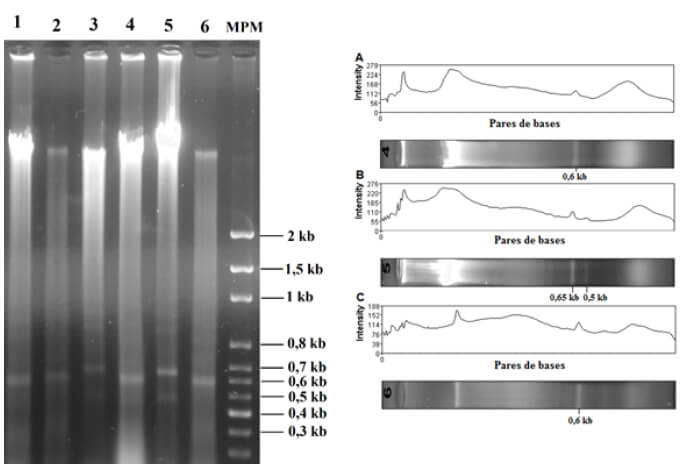
Por último, al evaluar la digestión con MspI, fue posible observar en el grupo control una banda de aproximadamente 0,6 kb, a diferencia del aislado resistente en donde se observan dos bandas: una correspondiente a 0,65 kb y otra de 0,5 kb. En el caso de la muestra proveniente de P1, se observa una banda única de 0,6 kb. Por lo tanto, fue posible identificar un patrón de restricción distinto en el aislado con resistencia inducida experimentalmente y el aislado proveniente del paciente con falla terapéutica.
En este trabajo se reportaron variaciones genéticas evidentes mediante el análisis de polimorfismos de longitud de fragmentos de restricción en el ADNt de aislados de Leishmania (V.) braziliensis, al comparar los patrones de restricción del ADNt de los parásitos aislados de pacientes (con y sin curación clínica), respecto a los aislados provenientes de cepas de referencia internacional (MHOM/BR/00/M2903) cuya resistencia fue inducida experimentalmente mediante inyecciones intra-lesionales en animales de experimentación y, posteriormente, con la presión selectiva del fármaco en medios de cultivo. En general, las bandas identificadas fueron de bajo peso molecular.
La diferencia entre los perfiles de restricción (con PstI o MspI) en los aislados de referencia (sensibles y resistentes) y el proveniente del paciente con falla terapéutica, apoya las hipótesis y conclusiones realizadas en estudios previos (Jeddi et al., 2011) (Adaui et al., 2011) que afirman el hecho de que aislados con resistencia in vitro, no necesariamente tienen la misma expresión genética que aquellos provenientes de pacientes sin curación clínica (es decir, con aparente resistencia). La expresión diferencial de ciertos genes implicados en el metabolismo de los antimoniales pentavalentes entre distintos aislados de L. (V.) braziliensis, hizo sugerir a Adaui y colaboradores en el 2011 (Adaui et al., 2011) una posible respuesta pleiotrópica del parásito ante la presión selectiva del fármaco, un hecho anteriormente evidenciado en L. donovani. Esto sugiere que, experimentalmente, es posible obtener respuestas diferentes durante la exposición al fármaco y con la misma especie, aun en condiciones controladas. Además, el hecho de que en este trabajo se haya encontrado un perfil ligeramente distinto en el paciente sin cura clínica sugiere pensar en la influencia de la localización geográfica y las características específicas del hospedero sobre el parásito, teniendo en cuenta plasticidad genética de Leishmania como género (Laffitte et al., 2016), provocando cambios epigenéticos (Rodríguez et al., 2002) que los diferencien, desde el punto de vista molecular, de aquellos manipulados experimentalmente.
Además, las diferencias observadas al comparar a los pacientes sin curación clínica (P1 y P3), en los que se evidenciaron ligeras diferencias, pueden explicarse por varias razones. La primera de ellas es que, en el supuesto caso de que ambos aislados fuesen resistentes, se ha reportado una gran diversidad genética (Gómez et al., 2013) inclusive entre parásitos de la misma especie, lo cual explicaría las diferencias observadas como consecuencia del genoma original del parásito. Por otro lado, suponiendo que no hubiese variabilidad genética implicada, y que efectivamente ambos fuesen resistentes, las modificaciones genéticas dadas por la presión selectiva de la droga no siempre es igual, teniendo en cuenta los distintos factores que pueden provocar cambios epigenéticos en Leishmania (Afrin et al., 2019) en un mismo momento, los cuales suelen estar relacionados al hospedador. A pesar de ello, la identificación de estos factores no fue el objetivo en este estudio, aunque constituye un tópico de vital importancia para nuevas investigaciones.
En el caso específico de L. (V.) braziliensis, la identificación de variantes genéticas (sin tomar en cuenta la resistencia al tratamiento) ha sido reportada previamente en la literatura. En Venezuela, se detectaron tres grupos de parásitos según cambios moleculares evidenciados mediante digestión con enzimas de restricción e hibridación in situ con la sonda LbJ38 (Rodríguez et al., 1997). Estos cambios se relacionaron con la heterogeneidad de las bandas observadas, cada una con peso molecular entre 0,5 y 1,6 kb (Rodríguez et al., 1998). Es por eso que, en el presente trabajo, las diferencias entre los aislados de pacientes con falla terapéutica podrían explicarse por otros factores inherentes a la variabilidad genética propia del parásito, más allá de la probable relación con la resistencia a los antimoniales pentavalentes. La diferencia de vectores y reservorios, los aspectos geográficos y ecológicos son factores que influyen a nivel mundial en la diversidad molecular del parásito, independientemente de la exposición a un fármaco (Rodríguez et al., 1998).
La distribución geográfica de los pacientes en este estudio se relaciona directamente con la cercanía del IBJC (localizado en el Distrito Capital). Sin embargo, por lo menos en relación al estado Miranda, este fue reportado como el tercero con mayor número de casos de la enfermedad en el último estudio de epidemiología molecular en el año 2001 (Rodríguez et al., 1997), siendo superado solo por Lara y Mérida. En esta oportunidad, la mayoría de los aislados correspondieron a L. (V.) braziliensis (62,12%), seguido de L. (L.) mexicana (9,09%), siendo el resto de los resultados (28,79%) negativos. Puede que esta tendencia se siga manteniendo en la actualidad, aunque son necesarios nuevos estudios epidemiológicos en el área para corroborarlo.
Fue posible comprobar que la manipulación experimental realizada en este trabajo, con la que se propone una nueva metodología para la inducción de resistencia in vivo (en vez de la clásica inducción in vitro) es capaz de inducir cambios moleculares evidentes, y que el hecho de que se observara un perfil ligeramente distinto en los aislados provenientes de pacientes con falla terapéutica, corrobora el hecho de que se trata de un fenómeno multifactorial en el que la resistencia al tratamiento no es la única posibilidad a considerar.
La identificación de marcadores genéticos de resistencia es importante para identificar nuevos blancos terapéuticos, y, por consiguiente, para el desarrollo de nuevas alternativas de tratamiento. Sin embargo, para que los resultados observados en estudios de resistencia sean aplicables al contexto clínico, es necesario el desarrollo de herramientas que cumplan con una serie de condiciones (Sereno et al, 2019): (a) fácil de estandarizar, (b) ser económico, (c) fácil de manejar o realizar y (d) fácil de interpretar. Es por ello que el estudio constante de las alteraciones genéticas debe profundizarse, con el objetivo de permitir una diferencia significativa en el manejo clínico de los pacientes.
Con base en los objetivos planteados inicialmente, se detectaron perfiles de restricción distintos al comparar un aislado proveniente de paciente con falla terapéutica respecto a aislados de L. (V.) braziliensis de referencia internacional sensibles y resistentes al antimoniato de meglumina, además de encontrarse diferencias significativas al comparar los aislados provenientes de pacientes sin curación clínica entre sí. Sin embargo, el tamaño de la muestra es pequeño y hace falta continuar este estudio incluyendo un mayor número, a fin de obtener resultados más concluyentes. Además, se logró formular una nueva metodología para la inducción de resistencia en los parásitos, lo cual puede ser de gran utilidad para la obtención de marcadores genéticos de resistencia al Glucantime®, que puedan ser útiles para la indicación del tratamiento a seguir en el caso de la leishmaniasis cutánea localizada.
A Zoraya De Guglielmo, Rosmary Pacheco y Henry Oviedo, personal del laboratorio de ingeniería genética del IBJC por la asesoría y colaboración técnica y logística.