La Sección de Biohelmintiasis del Instituto de Medicina Tropical de la UCV ha dedicado más de 20 años de investigación en mejorar los métodos de inmunodiagnóstico de enfermedades parasitarias e infecciosas. Se han venido desarrollando y evaluando diferentes estrategias de búsqueda de biomoléculas de origen sintético, específicamente fragmentos de proteínas, sobre todo de parásitos, que puedan funcionar como antígenos y ser reconocidos por los anticuerpos. Dos de estas estrategias se conocen como Pepscan y MABA (Multiple Antigen Blot Assay). La primera consiste en sintetizar una biblioteca de péptidos (posibles antígenos) unidos a un soporte para posteriormente ser enfrentados a diversos fluidos fisiológicos (suero, saliva y orina) de pacientes y detectar anticuerpos contra un patógeno en particular. Los péptidos más antigénicos son posteriormente sintetizados en mayor cantidad para luego ser utilizados mediante la técnica de MABA. Esta técnica se basa en la adhesión de hasta 26 diferentes antígenos (péptidos) a una tira de nitrocelulosa la cual permite detectar o descartar simultáneamente hasta 26 patologías. Gracias a estas dos estrategias, en nuestro laboratorio se han podido identificar candidatos antigénicos o biomoléculas para ser empleados en el diagnóstico de la malaria, esquistosomiasis y hepatitis A, entre otras patologías. En las zonas tropicales y sub-tropicales las coinfecciones son comunes en la población, por lo que una práctica médica racional e integral requiere de conocer la historia actual o el contacto anterior de un paciente frente a varios patógenos importantes, de allí la importancia de aprovechar la visita a los servicios de saludpara descartar otras enfermedades que no necesariamente fueron el motivo de consulta y ofrecer un tratamiento oportuno.
Palabras clave: péptidos sintéticos; Pepscan; antígenos; anticuerpos; MABA, biohelmintiasis; Instituto de Medicina Tropical; Inmunodiagnóstico;enfermedades infecciosas y parasitarias.
The Biohelminthiasis Section of the Institute of Tropical Medicine (UCV) has devoted more than 20 years of research to improving methods of immunodiagnosis of parasitic and infectious diseases. Different strategies for the search of biomolecules of synthetic origin have been developed and evaluated, specifically fragments of proteins, especially of parasites, that can function as antigens and be recognized by the antibodies. Two of these strategies are known as Pepscan and MABA (Multiple Antigen Blot Assay). The first one consists of the synthesis of a library of peptides (possible antigens) attached to a support to subsequently be confronted with various physiological fluids (serum, saliva and urine) from patients and to detect antibodies against a particular pathogen. The more antigenic peptides are synthesized in greater quantity and then used in the MABA technique. This technique is based on the adhesion of up to 26 different antigens (peptides) to a strip of nitrocellulose which can detect out 26 pathologies simultaneously. Thanks to these two strategies, our laboratory has been able to identify antigenic candidates or biomolecules for the diagnosis of malaria, schistosomiasis, and hepatitis A among others. In tropical and subtropical areas, coinfections are common in the population, so a rational and comprehensive medical practice requires knowing the current history or previous contact of a patient with several important pathogens, hence the importance of taking advantage of the visit to the health services to rule out other diseases that were not necessarily the reason for consultation and provide timely treatment.
Key words: synthetic peptides; Pepscan; antigens; antibodies; MABA
[email protected], [email protected], [email protected], [email protected], [email protected], [email protected]
A María la picó un chipo hace un mes y acudió a la consulta del Instituto de Medicina Tropical con una referencia para diagnosticar Mal de Chagas. Se le tomó una muestra de sangre para aplicarle una prueba que puede diagnosticar una batería de 26 enfermedades a la vez. Una semana después se le entregó a María un resultado negativo para Chagas, pero se le sugirió confirmar la infección por toxoplasmosis y HIV, pues dio positivo para estas otras enfermedades. Con un solo pinchazo, con tan solo unos microlitros de su suero, se aprovechó para detectar o descartar 26 patologías simultáneamente.
¿Ficción?¿Es posible? Si bien el personaje de María es ficticio, no así lo que es capaz de hacer este método de diagnóstico, todavía en fase experimental, conocido como MABA (Noya y col. 1998; Noya y col. 2009) y desarrollado en el Instituto de Medicina Tropical de la Facultad de Medicina de la Universidad Central de Venezuela.
La metodología diagnóstica capaz de detectar simultáneamente múltiples enfermedades bien sean ellas de origen infeccioso, autoinmune y alérgico, empleando cantidades muy pequeñas de suero, plasma o saliva se describirá a continuación de forma sucinta, pero sin entrar en excesivos tecnicismos.
Para la identificación de biomoléculas que pudieran ser candidatas en la detección de ciertas enfermedades, se pueden utilizar varias técnicas. Entre ellas, están el Pepscan y el MABA, ensayos novedosos que usamos de rutina en la Sección de Biohelmintiasis del Instituto de Medicina Tropical de la Facultad de Medicina de la UCV.
¿MABA? ¿Pepscan? Un acrónimo y una palabra que juntas pueden llegar a convertirse en herramientas complementarias y poderosas para la identificación de biomarcadores que pudieran ser posteriormente utilizadas en el diagnóstico de enfermedades infecciosas y también como biomarcadores del grado de patología o de cura.
¿Cómo se realiza el diagnóstico de las diferentes patologías infecciosas o de otra índole? Se trata de la secuencia de procedimientos (clínico, epidemiológico y de laboratorio) que permiten identificar cualquier estado patológico de diferentes orígenes. Un buen diagnóstico es fundamental para suministrar tratamiento oportuno y adecuado a los pacientes. Este puede implicar diversos procedimientos dependiendo de la enfermedad en cuestión.
Dentro de los métodos de diagnóstico de laboratorio destacan las técnicas de inmunodiagnóstico, que permiten tanto la detección como la cuantificación de anticuerpos contra antígenos del patógeno en cuestión, pudiendo encontrarse en distintos fluidos biológicos (plasma, suero, saliva, líquido cefalorraquídeo u orina) (biomarcadores de infección). Otra forma de inmunodiagnóstico involucra la detección de antígenos circulantes que pueden hallarse en los distintos fluidos ya mencionados, por parte de anticuerpos (monoclonales o policlonales monoespecíficos).Un antígeno es cualquier biomolécula (usualmente de naturaleza proteica) que tiene la capacidad de ser reconocido por un anticuerpo (inmunoglobulinas) específico o por un receptor celular.
Desde hace más 20 años, nuestra Sección ha venido desarrollando y evaluando diferentes estrategias de búsqueda de biomoléculas de origen sintético, específicamente fragmentos de proteínas, sobre todo de parásitos, que puedan funcionar como antígenos y ser reconocidos por los anticuerpos.
La mayoría de los métodos de inmunodiagnóstico utilizados en la actualidad emplean proteínas recombinantes de organismos parasitarios o infecciosos. Una proteína recombinante es una proteína que ha sido expresada en otro organismo que no es el original y que le sirve de factoría.
Si bien la principal ventaja de utilizar la proteína recombinante es que se dispone de una “copia” de la proteína original, con frecuencia su síntesis resulta compleja, poco reproducible y costosa en insumos para su posterior purificación. En la Figura 1, se muestra de forma sucinta la secuencia de técnicas que se integran en cuatro etapas que son aplicadas en la SBH, con el fin obtener péptidos sintéticos con fines diagnósticos.
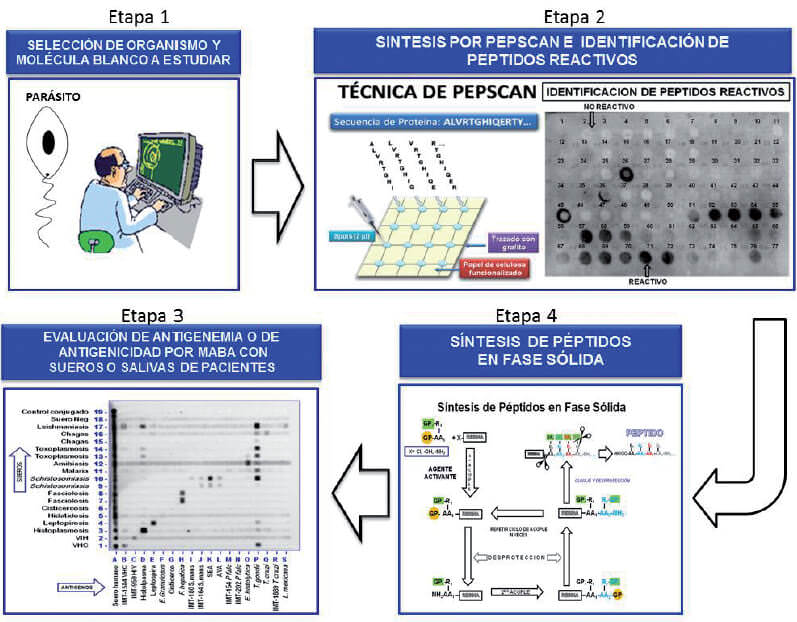
Muchas veces es innecesario preparar la proteína antigénica completa del parásito, ya que una sola porción de ella es necesaria para el reconocimiento por parte del anticuerpo, por lo que se recurre a la síntesis química de fragmentos de estas proteínas, llamados péptidos, que son moléculas más pequeñas que la proteína completa, casi siempre fáciles de sintetizar, a través de métodos reproducibles y con controles de calidad confiables. La porción del anticuerpo que reconoce el antígeno se conoce como epítopo.
Recientemente, la estrategia que hemos desarrollado y evaluado en nuestra Sección consta de cuatro etapas involucrando cada etapa varios pasos (Figura 1):
Etapa1. Estudio inmunoinformático para la selección de posibles secuencias antigénicas.
La finalidad del estudio inmunoinformático es identificar posibles antígenos o regiones antigénicas que puedan ser reconocidas por los anticuerpos. Las proteínas seleccionadas deberían cumplir dos requisitos: abundancia y antigenicidad. Para ello se procede así:
1.1. Identificación de una o más proteínas antigénicas: revisión de la literatura sobre moléculas ya reconocidas como antígenos o que al menos sus secuencias estén disponibles en las bases de datos del genoma/proteoma de los diferentes agentes causales.
1.2. Búsqueda en bases de datos información sobre la estructura primaria de los antígenos: en estas bases de datos se puede encontrar la estructura primaria total o parcial de la proteína, por ejemplo, la base de datos de proteínas del Centro Nacional de Información Biotecnológica (https://www.ncbi.nlm.nih.gov/).
1.3. Comparación de las estructuras primarias de las proteínas antigénicas versus proteínas semejantes de otros organismos: esto provee información que evite las posibles reacciones cruzadas con otros patógenos. Una reacción cruzada implica que otro patógeno diferente al que está bajo estudio presenta secuencias similares que son reconocidas por los sueros de pacientes con otra patología pudiendo conducir a un diagnóstico errado (“falso positivo”). Estas secuencias compartidas son descartadas mediante un algoritmo bioinformático conocido como herramienta de búsqueda de alineamiento local básico (siglas en inglés, BLAST) (Mount, 2007).
1.4. Determinación de antigenicidad: consiste en la selección de regiones antigénicas empleando servidores gratuitos disponibles en la web que utilizan distintos algoritmos de predicción. La antigenicidad es la propiedad del antígeno de ser reconocido, de manera específica, por un anticuerpo o un receptor celular.
Una vez determinadas las regiones antigénicas de la proteína del agente infeccioso, estas son sometidas un proceso de filtrado adicional utilizando los siguientes criterios:
1.4.1. Identificación de las regiones intrínsecamente desordenadas en la proteína: se da prioridad a epítopos localizados en regiones intrínsecamente desordenadas (o al azar). Estas regiones carecen de una estructura tridimensional bien definida y tienden a ser más antigénicas.
Una vez determinadas las regiones antigénicas de la proteína del agente infeccioso, estas son sometidas un proceso de filtrado adicional utilizando los siguientes criterios:
1.4.2. Determinación de secuencias en “tándem”: la presencia de repeticiones en “tándem” de una misma secuencia es otra de las características que contribuye a la antigenicidad e inmunogenicidad de una proteína. Las repeticiones en tándem son dos o más copias de la misma secuencia de aminoácidos.
1.4.3. Exclusión de regiones transmembranales y de péptidos señal: estas regiones corresponden a secuencias no expuestas insertadas dentro de la bicapa lipídica de las membranas y, por tanto, al no ser habitualmente antigénicas, se excluyen del estudio.
1.4.4. Estructura tridimensional del antígeno: si se dispone de información estructural (3D) de la proteína antigénica ésta debe ser considerada al momento del estudio, pues aquellas secuencias del exterior de las proteínas son habitualmente hidrofílicas y tienden a ser más antigénicas.
Llegado a este punto se dispone de un número considerable de secuencias candidatas a ser sintetizadas a fin de continuar con la etapa 2.
Una vez finalizado el estudio inmunoinformático y ya habiendo seleccionado las secuencias candidatas a ser evaluadas, se procede a la síntesis y evaluación de los posibles péptidos antigénicos seleccionados en la etapa 1 mediante la técnica Pepscan. Esta técnica consiste en la síntesis simultánea de varios péptidos sobre un soporte sólido, que no es más que un papel de filtro activado llamado membrana. El principal atractivo de esta técnica es que permite sintetizar más de 100 péptidos simultáneamente en forma de círculos o “spots” con la concentración suficiente de péptido para realizar posteriormente inmunoensayos e identificar a aquellos péptidos reactivos o inmunodominantes.
La técnica Pepscan (Geysen y col. 1984; Frank. 2002; Hilper y col. 2007) consiste en la síntesis de péptidos en forma de puntos (spots) unidos covalentemente sobre un soporte sólido (papel de filtro) funcionalizado que contiene grupos químicamente reactivos.
2.1. Preparación del soporte de celulosa: para hacer que la membrana de celulosa (papel de filtro) adecuada para la síntesis de péptidos es necesario modificar su superficie y cambiar la funcionalización de grupos hidroxi (-OH) a grupos amino (-NH2) siendo este último grupo más reactivo que el primero. El aminoácido protegido Fmoc-Gly-OH puede ser utilizado para la funcionalización del papel de filtro.
La modificación de la membrana de celulosa también involucra la inserción de una molécula que sirve de espaciador permitiendo mejorar la accesibilidad de los grupos –NH2 de la membrana. Este espaciador incrementa la distancia del péptido sintetizado desde la superficie de la membrana mejorando la accesibilidad de los anticuerpos durante los ensayos biológicos. Un ejemplo de espaciador que está siendo utilizado por nosotros es el dipéptido o tripéptido SerGly o GlySerGly respectivamente. Previo a la funcionalización se debe recortar el papel de filtro del tamaño deseado y cuadricular con un lápiz de grafito. Es en la intersección de las líneas en la cuadricula el lugar donde se sintetizará cada spot.
2.2. Síntesis de los péptidos: una vez funcionalizada la membrana, se procede a la síntesis de cada péptido sobre la membrana en forma de círculos o spots. Los péptidos son sintetizados sobre la membrana utilizando la estrategia fluorenilmetoxicarbonil (Fmoc) (Atherton y col. 1978) empleando aminoácidos protegidos en el grupo –NH2 con el grupo protector Fmoc (protección temporal) y las cadenas laterales de los aminoácidos también protegidas (protección permanente) para evitar reacciones colaterales durante el proceso de elongación de la cadena peptídica.
De forma muy general, el grupo funcional ácido carboxílico del aminoácido protegido es activado químicamente utilizando un agente acoplante para luego realizar una reacción de condensación con los grupos –NH2 presentes en la membrana, formando el enlace peptídico. Este paso se conoce como acoplamiento. Una vez incorporado el primer aminoácido, se debe eliminar la protección temporal Fmoc de este para poder acoplar un segundo aminoácido. Este paso de denomina desprotección. Una vez realizado esto, se procede a la activación del segundo aminoácido. El proceso se repite hasta sintetizar el péptido con el número de residuos de aminoácido deseado. Por lo general, todos los péptidos sintetizados en el Pepscan tienen el mismo número de residuos de aminoácidos, pero pueden sintetizarse de tamaños variados.
Cada reacción de desprotección y acoplamiento es monitoreada utilizando un indicador ácido-base llamado azul de bromofenol.
Luego del último acople, se procede a la desprotección de los grupos permanentes presentes en el péptido sintetizado, utilizando un ácido fuerte tal como el ácido trifluoroácético y una serie de “scavengers” (“atrapadores”) que permiten atrapar estos grupos protectores liberados, evitando que se unan a la cadena peptídica y alteren la estructura química del péptido.
En este punto, cada péptido permanece anclado a la membrana de celulosa y está lista para ser utilizada en los inmunoensayos.
2.3. Determinación de antigenicidad de los péptidos: una vez expuesto el papel con los péptidos a los anticuerpos y revelado, permite identificar los spots reactivos, que serán seleccionados para su posterior síntesis en fase sólida, y su evaluación por MABA o por una técnica de ELISA convencional frente a sueros seleccionados u otros fluidos corporales a estudiar. La exposición secuencial de los péptidos a los anticuerpos presentes en la muestra de los pacientes, seguido por el uso de anticuerpos antihumanos conjugados a una enzima, que en presencia de un sustrato cromogénico o luminiscente, dará en el caso de aquellos péptidos reactivos una señal de color o luminosa respectivamente en forma de mancha circular o “spots” (Figura 1, etapa 2). Ese mismo papel puede ser reutilizado hasta cuatro veces con diferentes anticuerpos, siempre y cuando los anticuerpos de los sueros anteriores sean removidos con una solución conteniendo urea y SDS (Härkönen y col. 2002; Kovacs-Nolan y col. 2003).
Los péptidos que sean reconocidos durante el inmunoensayo del Pepscan por los anticuerpos de los pacientes, son seleccionados para ser sintetizados en mayor cantidad mediante dos posibles metodologías: t-BOC (Houghten y col. 1986) y Fmoc (Atherton y col. 1978), que permiten sintetizar péptidos en fase sólida y el producto resultante tiene el aspecto de polvo cristalino generalmente de color blanco, que podrá ser evaluado por diferentes técnicas inmunológicas, tales como las técnicas de ELISA (Voller, 1978) y de MABA (Noya y Alarcón de Noya, 1998). Una vez sintetizados los péptidos se procede a su control de calidad evaluando el peso molecular de cada péptido mediante espectrometría de masas (EM) y determinando su pureza mediante cromatografía líquida de alta eficiencia (HPLC).
El mapeo previo mediante la técnica de Pepscan permite seleccionar los péptidos más relevantes y reducir el número de ellos para luego evaluarlos por MABA. Esta prueba permite estudiar una gran cantidad de sueros de pacientes en un solo experimento y comprobar cómo funcionan los péptidos como candidatos para el inmunodiagnóstico masivo. Su mayor ventaja se basa en la evaluación comparativa rápida, económica y simultánea de varias preparaciones antigénicas frente a muchos sueros, permitiendo el multidiagnóstico simultáneo de una gran variedad de enfermedades infecciosas y parasitarias.
A continuación, se describe de forma muy general, en qué consiste esta técnica.
4.1. Técnica MABA: el acrónimo MABA, corresponde a las siglas del inglés Multiple Antigen Blot Assay. Es una técnica “hecha en casa” diseñada en nuestro laboratorio (Noya y Noya, 1998, Noya y col. 2009), versátil, confiable, reproducible y económica para el análisis de múltiples compuestos de manera simultánea. Se basa en la adhesión de hasta 26 diferentes antígenos (en este caso péptido) a una tira de nitrocelulosa usando un dispositivo acrílico de origen industrial y comercial (Miniblotter®) con canales paralelos.
4.1.1. Utilizando este dispositivo acrílico, se sensibiliza un papel de nitrocelulosa (NC) u otro soporte de similares características con los diferentes péptidos a estudiar a través de canales paralelos.
4.1.2. Bloqueo de la membrana de NC: una vez se adhieren los péptidos al papel de NC, se bloquean las áreas sin péptidos con leche descremada para evitar la adhesión inespecífica de los anticuerpos y luego se corta de forma perpendicular a los canales, de manera que todos los compuestos queden alineados como recuadros antigénicos en una sola tira.
4.1.3. Análisis de antigenicidad de los péptidos por los sueros: las tiras de NC conteniendo los recuadros de la hilera de diferentes péptidos, se incuban individualmente con sueros (u otros fluidos) de pacientes infectados. Posteriormente se revela si hubo reconocimiento con un anti-anticuerpo humano marcado con una enzima y el conjugado apropiado para comprobar si los anticuerpos de la muestra reaccionan con alguno(s) de los compuestos (antígenos) adsorbidos a la membrana de NC.
4.1.4. Revelado del reconocimiento anticuerpospéptidos: las señales positivas son registradas usando sustratos quimioluminiscentes o colorimétricos precipitables, tal como fuera señalado en el punto 2.3.
Con esta estrategia, es posible determinar el perfil de enfermedades infecciosas (de exposición presente o pasada) mediante la detección de anticuerpos contra una o más enfermedades, lo cual debe ser luego confirmado con pruebas especializadas para cada una. De esta manera, unos pocos microlitros de sangre (60 microlitros) o saliva pueden ser útiles para detectar simultáneamente hasta 26 enfermedades.
El MABA permite la demostración cualitativa simultánea de la antigenicidad e inmunogenicidad de antígenos obtenidos en forma de péptidos sintéticos, moléculas recombinantes o preparaciones crudas, con alta sensibilidad y especificidad y bajo costo.
Algunas variantes y mejoras, como el Cross-MABA permiten hacer una evaluación cuantitativa del grado de reacción de cada suero (Noya y col. 2015). Otra ventaja del MABA es que un único ensayo permite la evaluación de al menos 26 sueros diferentes contra 26 antígenos diferentes, lo cual es equivalente a 8 placas de ELISA convencionales. El costo por evaluación/antígeno/paciente es de 0.006$ por MABA vs. 0.129 $ por ELISA.
Cabe destacar que esta es una de las diversas estrategias que pueden llevarse a cabo para identificar péptidos sintéticos de valor como biomarcadores de infección, patología y cura. Su éxito dependerá en primer lugar de cuánta información se disponga del proteoma del patógeno, ya que no todas las proteínas caracterizadas en el proteoma son antigénicas o inmunogénicas.
Recientemente nuestro equipo ha identificado, bajo la estrategia anteriormente expuesta, nuevos antígenos basados en péptidos sintéticos para el diagnóstico de Hepatitis A mediante Pepscan, MABA y ELISA: Un péptido dímerico identificado como IMT-1996, localizado en el extremo C-terminal de la proteína VP1, resultó antigénico con una frecuencia de reconocimiento de anticuerpos anti-IgG entre el 87–100% y 100% de reconocimiento para anticuerpos anti-IgM (Gauna y col. 2015).
Asimismo, hemos utilizado la técnica del Pepscan para seleccionar epítopos relevantes para el diagnóstico de hepatitis C, esquistosomiasis y malaria (Zerpa y col. 2006). Luego, mediante el MABA ha sido posible evaluar los epítopos escogidos frente a un gran número de sueros de pacientes, con lo cual hemos obtenido frecuencias de reconocimiento suficientemente altas para algunos de ellos y por lo tanto se han usado en el tamizaje preliminar de poblaciones para luego ser confirmados por pruebas particulares en cada caso. Por ejemplo, péptidos derivados de la proteína Sm31 de Schistosoma mansoni fueron reconocidos por 49-89% de pacientes con bilharzia, con 100% de especificidad (Noya y Noya, 1998). También, mediante MABA se identificaron péptidos inmunodominantes de la proteína Sm32 de Schistosoma mansoni (Noya y col. 2003).
De esta manera, se detectan anticuerpos en sueros de los pacientes, así como adicionalmente se puede evaluar la inmunogenicidad de estos epítopos en ratones, conejos o gallinas para producir anticuerpos policlonales monoespecíficos capaces de reconocer esas moléculas en ensayos de captura de antígenos circulantes en fluídos corporales.
El MABA puede ser implementado y adaptado para poblaciones e instituciones particulares, tales como bancos de sangre y laboratorios clínicos, así como para detectar enfermedades en pacientes pediátricos y en inmunocomprometidos. También ha sido útil en estudios de reactividad cruzada (Losada y col. 2005) y en la evaluación de protocolos de vacunación (Noya y col. 2007, Noya y col. 2010, Chacón y col. 2003, Sulbaran y col. 2013), siendo estas otras de las líneas de investigación importantes de nuestro laboratorio.
En estos momentos ambas técnicas están siendo utilizadas en la búsqueda de biomoléculas que pudieran ser empleadas en el diagnóstico de la malaria usando saliva, con resultados alentadores.
Finalmente, es importante recordar que las coinfecciones son muy comunes en los países en desarrollo, por lo que una práctica médica racional e integral requiere de conocer la historia actual o el contacto anterior de un paciente frente a varios patógenos importantes, de allí la importancia de aprovechar la visita a los servicios de salud para descartar otras enfermedades que no necesariamente fueron el motivo de consulta y ofrecer un tratamiento oportuno.