El proyecto genoma de Trypanosomacruzi reveló la existencia de la segunda familia más grande de genes de este parásito causante de la enfermedad de Chagas: la familia MASP. Las proteínas MASP son antigénicas por lo que nos propusimos expresar y purificar una proteína MASP recombinante para una posterior caracterización inmunológica con sueros chagásicos venezolanos. Con este fin se realizó un despistaje inmunológico con sueros de pacientes chagásicos venezolanos, de una genoteca de genes MASP construida mediante amplificación por PCR con cebadores específicos de las regiones 5' y 3' conservadas y subsecuente clonación en el vector de expresión pT7-MAT-2, que contiene una etiqueta de histidinas. Luego de despistajes primario y secundario se seleccionó el clon CM82 que mostró un inserto completo de 400 pb. Se indujo la expresión de la proteína CM82 a partir de un cultivo de bacterias E. coli incubadas con IPTG durante 4 horas y se purificó la proteína recombinante por cromatografía de afinidad a metal inmovilizado. La electroforesis en gel de poliacrilamida-SDS, reveló una proteína purificada de aproximadamente 28 kDa. El ensayo de DOTELISA, usando una mezcla de suero de pacientes chagásicos, permitió verificar su antigenicidad.
Palabras clave: MASP; Trypanosomacruzi; Trypanosomarangeli; antígeno recombinante; Chagas; purificación
The genome project of Trypanosoma cruzi revealed the existence of the second largest family of genes of this parasite that causes Chagas disease: the MASP family. The MASP proteins are antigenic, so we proposed to express and purify a recombinant MASP protein for subsequent immunological characterization with venezuelanchagasic sera. To this end, immunological screening with sera from venezuelanchagasic patients was performed from a MASP gene library constructed by PCR amplification with specific primers from the conserved 5 'and 3' regions and subsequent cloning into the expression vector pT7-MAT-2, Which contains a histidine tag. After primary and secondary screenings clone CM82 was selected which showed a complete 400 bp insert. Expression of the CM82 protein was induced from a culture of E. coli bacteria incubated with IPTG for 4 hours and the recombinant protein was purified by immobilized metal affinity chromatography. SDS-polyacrylamide gel electrophoresis revealed a purified protein of approximately 28 kDa. The DOTELISA assay, using a serum pool of chagasic patients, allowed to verify its antigenicity.
Key words: MASP, Trypanosomacruzi; Trypanosomarangeli; recombinantantigen; Chagas; purification
Trypanosomacruzi, el agente etiológico de la enfermedad de Chagas posee un genoma con un 50% de secuencias repetidas, las cuales están organizadas en grandes familias de genes de proteínas de superficie, retrotransposones y repeticiones subteloméricas.
Las proteínas de superficie asociadas a mucinas, llamadas MASP por sus siglas en inglés, constituyen la segunda familia más grande de este parásito. Fueron inicialmente descritas a raíz de la secuenciación del genoma de T. cruzi en 2005 (El-Sayed et al. 2005), siendo por primera vez demostrada su transcripción en T. cruzi por Abate et al. ese mismo año (Abate et al. 2005).
La familia MASP está caracterizada por dominios altamente conservados en los extremos N y C terminales y una región central variable. Compone al menos el 6% del genoma diploide de T. cruzi y debe su nombre al hecho de estar ubicada entre los genes que codifican para las trans-sialidasas y mucinas. En concreto, aguas abajo de los miembros de TcMUC II con los cuales se asemejan en estructura no así en secuencia (El Sayed et al. 2005).
Las proteínas MASP se expresan preferencialmente en tripomastigotes. Poseen sitios para la N y O-glicosilación, sufriendo extensas modificaciones postraduccionales (Atwood y et al. 2005). Estas proteínas se han asociado a la virulencia del parásito, participando en procesos de evasión del sistema inmune e invasión celular (De Pablos et al. 2016). Algunas proteínas MASP se secretan, ya sea individualmente o mediante exovesículas, mientras que otras permanecen unidas a la membrana del parásito. La expresión de una determinada MASP está limitada a un grupo de la población total de parásitos (Bartholomeu et al. 2009). Se ha confirmado la existencia de una respuesta inmune humoral en el suero de pacientes chagásicos hacia el segmento conservado C-terminal de los miembros de la familia MASP (De Pablos et al. 2016) quienes determinaron que la secuencia DGSTAV es el epítope más inmunogénico de la región C-terminal.
El diagnóstico inmunológico de la enfermedad de Chagas se realiza usando principalmente la técnica de ELISA (Luquetti et al. 2010). Esta técnica ha demostrado una alta especificidad y sensibilidad, dependiendo del antígeno que se use, entre los cuales están proteínas recombinantes y extractos del parásito crudos o semipurificados (Duarte et al. 2014). Sin embargo, aún no existe un antígeno que haya sido aceptado como reactivo de referencia para un diagnóstico unificado, debido en parte al diferente desempeño que demuestran entre los diversos estudios reportados. En este contexto, el presente trabajo reporta la clonación, expresión y purificación de una proteína MASP recombinante como método que podría conducir a la producción de un antígeno útil para ser evaluado en el diagnóstico inmunológico de la enfermedad de Chagas.
La cepa de Escherichia coli empleada para el clonamiento y la expresión fue BL21(DE3), cuyo genotipo es huA2 [lon] ompT gal (λ DE3) [dcm] ∆hsdS λ DE3 = λ sBamHIo ∆EcoRI-B int::(lacI::PlacUV5::T7gene1) i21 ∆nin5 (Studier y Moffat, 1986).
Los epimastigotes de la cepa EP de Trypanosoma cruzi (un aislado humano venezolano) se cultivaron en medio LIT a 28ºC (Camargo 1964). Para el control positivo en los ensayos inmunológicos se utilizó como antígeno un extracto crudo de epimastigotes de esta cepa (Maekelt y Colmenares De Alayón 1960) deslipidado y liofilizado para almacenamiento a -20°C (Díaz-Bello et al. 2008), con una concentración de 1mg/mL luego de su reconstitución enagua destilada.
Para los ensayos inmunológicos se utilizó una mezcla de sueros de pacientes con enfermedad de Chagas (n = 16) diagnosticados por un ensayo de ELISA con extracto crudo antigénico de epimastigotes de T. cruzi (Maekelt y Colmenares De Alayón 1960) y por hemaglutinación indirecta. Como control negativo se usó una mezcla de sueros de individuos sanos (n = 20) confirmados como negativos por los dos ensayos serológicos ya mencionados.
Todos los sueros provienen de la Sección de Inmunología del Instituto de Medicina Tropical, Universidad Central de Venezuela, Caracas, Venezuela.
Para la utilización de estos sueros se contó con el consentimiento previa información de los pacientes e individuos sanos y con el aval de la Comisión de Bioética del Instituto de Medicina Tropical de la Universidad Central de Venezuela.
La genoteca se construyó mediante amplificación por PCR de la familia de genes MASP usando ADN genómico de epimastigotes de la cepa EP de T. cruzi purificado con DNAZOL (Thermo Fisher Scientific, USA), de acuerdo con las instrucciones de la casa comercial.
Para la PCR se diseñaron los cebadores FXhoI (5’CGCGGCCTCGAGATGGCGATGATGATGACG 3’) y RBglII(5’ – GATCTAGAAGATCTTTCGCGGCCACCACCGC-3´) específicos de las regiones 5' y 3' conservadas, comunes a todos los miembros de la familia de genes MASP. Estos cebadores contienen los sitios de restricción XhoI y BglII, que permitieron la inserción de los productos de amplificación en el vector de expresión pT7-MAT-2 (SIGMA, USA) que contiene una etiqueta de histidina. La PCR se llevó a cabo en un volumen final de 25 ul que contenía MgCl2 1,5 mM,dNTP 200 µM, GoTaq Hot StartPolymerase (Promega, USA) 0,5 U, cebador FXhoI 0,2 µM, cebador RBglII 0,2 µM, tampón 1X. Las condiciones de ciclado fueron: desnaturalización a 95°C durante 1 min, 30 ciclos de 95°C por 1 min, 60°C por 1 min, 72°C por 1 min y una extensión final a 72°C por 5 min. Los productos de amplificación (genes MASP), así como el vector pT7-MAT-2 se digirieron con las enzimas de restricción XhoI y BglII (Promega, USA) de acuerdo a las instrucciones de la casa comercial. Una vez digeridos se purificaron con el estuche PureLink™ PCRPurification Kit (Thermo Fisher Scientific, USA) siguiendo las instrucciones de la casa comercial y se cuantificó por espectrofotometría en un espectrofotómetro SmartSpect 3000 de BIO-RAD. A continuación, se realizó la ligación inserto-vector con el estuche Ligase T4 Rapid DNA Ligation Kit (Thermo Fisher Scientific, USA), siguiendo sus instrucciones y con esta mezcla de ligación se transformaron bacterias E. coli BL21 competentes (Ausubel et al. 2003). Las bacterias transformantes, que constituyen la genoteca de genes MASP, se seleccionaron por crecimiento en medio selectivo con ampicilina, marcador de resistencia del vector pT7-MAT-2.
A partir de las placas de LB agar con las colonias transformantes de la genoteca se hicieron réplicas en membrana de nitrocelulosa que fueron colocadas en nuevas placas de LB agar suplementado con IPTG 0,5 mM e incubadas durante 4 horas a 37°C. Las colonias crecidas se lisaron colocando la membrana de nitrocelulosa en una atmósfera saturada de cloroformo durante 10 min e incubándolas a continuación en una solución de lisis (Tris-HCl 25 mM, EDTA 10 mM pH 8.0, lisozima 200 ng/mL) durante 1 h a temperatura ambiente. Se lavaron las membranas 2 veces por 10 min cada vez en TBST (Tris-HCl 20 mM, pH 7.5, NaCl 150 mM, Tween 20 0,5%) y se transfirieron a una solución de bloqueo (leche descremada 5% en TBST) a 4°C durante la noche. Las membranas se incubaron en 10 ml de una mezcla de sueros de pacientes chagásicos diluidos 1:250 que contenía 500 ul de lisado de E. coli BL21, durante 1 h a temperatura ambiente. Se hicieron 4 lavados de 15 minen TBST y se incubaron 1 h con anti-IgG humana de cabra conjugada con peroxidasa de rábano picante (Thermo Fisher Scientific, USA). Luego se realizaron 4 lavados en TBST y 1 en TBS, para entonces incubar con la solución de sustrato quimioluminiscente (SuperSignal West Pico ChemiluminescentSubstrate, Pierce, USA) y detectar la señal en un Chemidoc Imagen System (BIO-RAD, USA).
A las colonias que resultaron positivas se les realizó un despistaje inmunológico secundario siguiendo el mismo procedimiento.
Una de las colonias positivas en el despistaje inmunológico, que denominamos CM82, se cultivó en caldo LB suplementado con ampicilina durante la noche a 37°C, para inocular al día siguiente 250 ml de LBhasta alcanzar una DO600= 0,6 U. En ese momento se añadió IPTG 0,5 mM para inducir la expresión durante 4 h a 37°C. Luego de una centrifugación a 3000 x g, por 10 min a 4°C, se resuspendió el sedimento en 10 ml de solución de lisis del estuche QiaEXPRESS Ni-NTA (Qiagen, Alemania) y se purificó la proteína recombinante a partir de la fracción soluble del lisado bacteriano por cromatografía de afinidad a metal inmovilizado siguiendo las instrucciones de la casa comercial. Se analizó un volumen de 5 ul de cada fracción de la purificación en un gel de poliacrilamida-SDS al 17% (Laemmli 1970). La cuantificación de proteínas de hizo siguiendo el protocolo descrito por Bradford (1976).
Se aplicaron 1, 5 y 10 ul del eluato con la proteína recombinante CM82 purificada, así como de los eluatos provenientes de cultivos de bacterias con el plásmido pT7-MAT-2 sin inserto (usado como control negativo), a una membrana de nitrocelulosa que fue entonces procesada para la detección inmunológica con mezcla de sueros de pacientes chagásicos tal como se describió para el despistaje inmunológico de la genoteca.
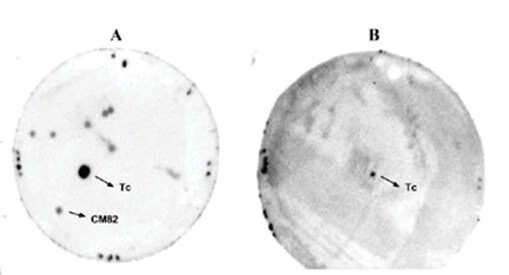
La clonación de un gen de la familia MASP de T. cruzi codificante de una proteína con actividad antigénica se llevó a cabo mediante el despistaje inmunológico con sueros de pacientes chagásicos, de una genoteca construida por amplificación PCR usando cebadores específicos de las regiones 5' y 3' conservadas comunes a todos los miembros de esta familia de genes. Luego de despistajes primario y secundario se obtuvo un clon que llamamos CM82 (Fig. 1).
El crecimiento de CM82 en presencia de IPTG indujo la expresión de la proteína MASP-CM82 recombinante, que fue entonces purificada por cromatografía de afinidad a metal inmovilizado, utilizando la etiqueta de histidina presente en el vector de expresión pT7-MAT-2 usado para la construcción de la genoteca. La Fig. 2 muestra una banda de aproximadamente 25 kDaeluida de la columna de purificación (carril 6). Usando el método de Bradford no se logró cuantificar la cantidad de proteína purificada, presumiblemente por estar bajo el límite de detección del ensayo.
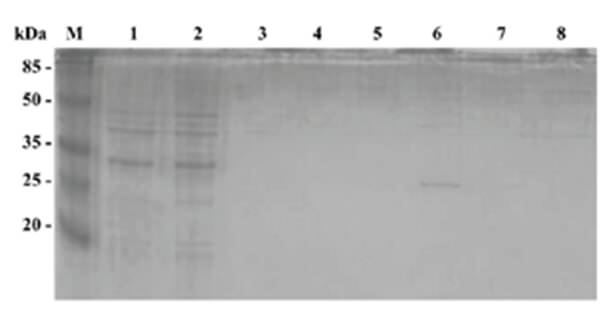
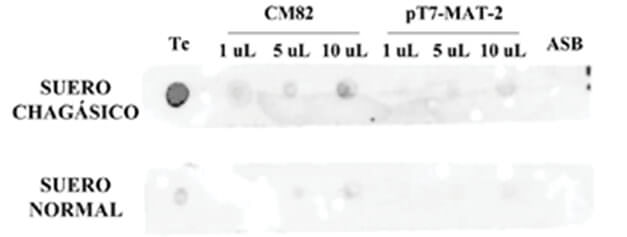
Cuando se aplicó a la columna el sobrenadante del lisado de bacterias E. coli BL21 con el vector pT7-MAT-2 sin inserto e inducidas con IPTG, usadas como control negativo de todo el procedimiento, no se observó purificación de ninguna proteína (carriles 5 y 7).El ensayo de DOT-ELISA, usando una mezcla de suero de pacientes chagásicos, permitió verificar la antigenicidad de la proteína recombinante CM82, tal como se muestra en la Fig. 3.
Recientemente se demostró la presencia de una respuesta inmune humoral de las proteínas MASP de T. cruzi en humanos (De Pablos et al. 2016). Los autores usaron como antígeno, 6 péptidos sintéticos solapados correspondientes a la secuencia consenso de la región C-terminal de estas proteínas, demostrando un reconocimiento específico de anticuerpos hacia esta región. Esta antigenicidad convierte a las proteínas MASP en candidatos ideales para el desarrollo de ensayos diagnósticos serológicos que puedan complementar y/o mejorar los ya existentes.
En el presente trabajo reportamos la obtención de una proteína MASP recombinante purificada que es reconocida por sueros de pacientes chagásicos venezolanos, como primer paso para su evaluación inmunológica en ensayos diagnósticos.
La producción de proteínas recombinantes en sistemas bacterianos es una opción simple y económica para la obtención de cantidades suficientes de la proteína de interés.
Con el fin de asegurar la antigenicidad de la proteína expresada por el clon a utilizar para purificar la MASP recombinante, se realizó despistaje inmunológico de una genoteca de expresión de genes MASP con sueros de pacientes chagásicos.Es así como, efectivamente, se logró seleccionar un clon reactivo a partir del cual se indujo la expresión de la proteína recombinante y se purificó por afinidad en columna de metal inmovilizado.
Sin embargo, el rendimiento de la purificación fue muy bajo, ya que la cantidad de proteína en el eluato estuvo por debajo del límite de detección del método de Bradfod, que en nuestro caso fue 125 ug/mL. A pesar de ello, sí fue posible verificar su antigenicidad específica en un ensayo de DOT-ELISA que es ideal, por su simplicidad y costos relativos, para ser adaptado a laboratorios clínicos.
En conclusión, logramos implementar un método para la obtención de proteínas MASP recombinantes que podrían ser usadas en la evaluación de nuevos ensayos para el diagnóstico serológico de la enfermedad de Chagas.
Este trabajo fue financiado por FONACIT, Ministerio del Poder Popular para la Ciencia y Tecnología (MPPCT) Proyecto 2007001425 y por Consejo de Desarrollo Científico y Humanístico, UCV, Proyecto 20060000216.